Cientista de dados, aquele ser que pode ser imaginado como um super-geek por trás dos modelos solucionadores de mistérios. Bom isso acontece, mas o que pouca gente conta é que um bom pedaço do tempo desse profissional é investido em dataPrep, nada cool, costuma ser demorado e delicado, essa última parte quer dizer que “não, não da para automatizar 100% do dataPrep, o ideal é sempre ter um humano se certificando de que tudo está bem”. É inevitável analisar alguns histogramas, estudar dados faltantes, fazer filtros, agrupamentos e tudo o mais.
É aí que entra o data.table. Ah, data.table! Essa belezinha simplesmente é o jeito mais legal de virar sua base de dados ao avesso de forma “braçal”, é uma biblioteca que permite ao usuário adotar uma ‘linguagem’ de manipulção de base que é curta e, na grande maioria das vezes, bem mais rápida que a concorrência.
Ok, ok, isso é fala de fã, admito. O que vou fazer aqui então é dar uma pequena demonstração de como um comando pode ser passado do aclamado dplyr para datatable de forma simples, uma vez que se entende a estrutura, e como isso faz diferença no desempenho do script. Para isso vamos começar construindo uma base de 20 milhões de linhas e 10 colunas.
library(data.table)
library(dplyr)
library(purrr) # For map() function
library(microbenchmark) # For benchmarking
library(tidyverse)
dfs <- rep(2e7,10)
set.seed(123456)
myList <- map(dfs, #amostra de 20kk de valores no range de 1:1e5, 10 vezes
base::sample,
x = 1:1e5,
replace = TRUE) %>%
map(matrix, # em cada posicao da lista, quebra o vetor em 10 e joga cada parte como coluna de uma matriz
ncol = 10) %>%
map(data.frame) # transforma todas matrizes da lista em data.frames
df3 <- data.table::rbindlist(myList) # une as bases na lista
Antes de prosseguir, vamos dar uma olhada na base.
df3
X1 X2 X3 X4 X5 X6 X7 X8 X9 X10
1: 87921 55793 81282 39316 43389 54449 60047 18947 21528 46857
2: 78535 91446 36566 17892 6147 33818 67607 22926 26595 30526
3: 6326 76229 60797 92675 1069 23344 51051 30793 47039 52544
4: 76518 66161 48164 35810 32729 87865 21972 73380 88603 11411
5: 57728 2418 17606 59877 72380 39148 85925 4679 824 28162
---
19999996: 61381 43589 70131 41942 32774 19965 4749 52238 50814 50884
19999997: 45927 4094 38977 76729 90735 21795 9222 63427 70221 26999
19999998: 99080 70908 9367 14237 39944 77173 36196 95234 31328 47128
19999999: 60724 37080 4661 49043 70521 87968 38904 10466 16827 46864
20000000: 4670 63739 27229 73348 72747 21393 29047 91010 44935 96431
Agora suponha que estamos na fase de dataPrep desta base, tentando descobrir coisas sobre ela como : “ Qual o valor minimo de X3 agrupado por X1?”. Vamos escrever isso em dplyr e datatable para entender como é simples traduzir de um para o outro.
# escrevendo em datatable
teste1 <- df3[,.(min_X3 = min(X3)),.(X1)][order(X1)]
# escrevendo em dplyr
teste2 <- df3 %>%
group_by(X1) %>%
summarize(min_X3 = min(X3))
# conferindo se as saídas são iguais
all.equal(teste1,as.data.table(teste2))
[1] TRUE
Detalhando o que acontece no código acima, partindo do entendimento de dplyr
output <- data %>% # primeiro passo: Fornecer a base a ser manipulada
group_by(var) %>% # segundo passo: informar variável de agrupamento
summarize( var_label = func(var2)) # terceiro passo: aplicar função na variável de interesse, dando resultado por grupo.
# A tradução para datatable pode ser confusa no princípio porque apesar de ter uma ordem cronológica, no datatable talvez essa ordem não seja tão intuitiva.
# estrutura básica para operar com datatable
data[filtro, .(summarize), .(group_by)]
# para entender ideia de ordem no datatable temos que:
data[ filtro passo 1, .(summarize passo 1), .(group_by passo 1)]
# para ir ao passo 2 teríamos
data2 <- data[ filtro passo 1, .(summarize passo 1), .(group_by passo 1)]
data2[ filtro passo 2, .(summarize passo 2), .(group_by passo 2)]
# mas a beleza do pacote é fazer com que você precise enderessar objetos o mínimo possível então da para escrever assim
output <- data[ filtro passo 1, .(summarize passo 1), .(group_by passo 1)][ filtro passo 2, .(summarize passo 2), .(group_by passo 2)] ... [ filtro passo n, .(summarize passo n), .(group_by passo n)]
#vale lembrar que é por conta da estrutura básica que quando declaramos teste1, precisamos começar com vírgula, para indicar que não queremos filtros no nosso cálculo.
#Fora isso basta copiar o que você colocaria no 'summarize' para o espaço após a primeira virgula e copiar o que você colocaria no 'group_by' após a segunda vírgula.
Agora que já sabemos como gerar a mesma operação por grupo nos dois pacotes, vamos comparar o desempenho com microbenchmark, esta função executa uma expressão um dado número de vezes e conta o tempo gasto em cada iteração. Para esse caso, foram feitas 100 execuções de cada expressão.
time_champs <- microbenchmark({
df3[,.(min_X3 = min(X3)),.(X1)][order(X1)]
}, times = 100L)
time_nonchamps <- microbenchmark({
df3 %>%
group_by(X1) %>%
summarize(min_X3 = min(X3))
}, times = 100L)
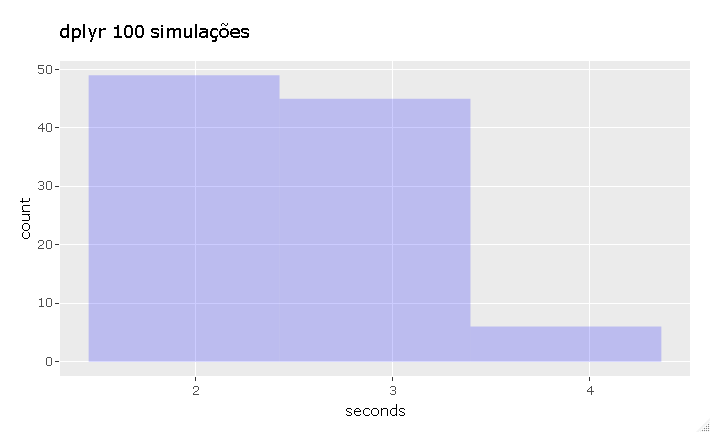
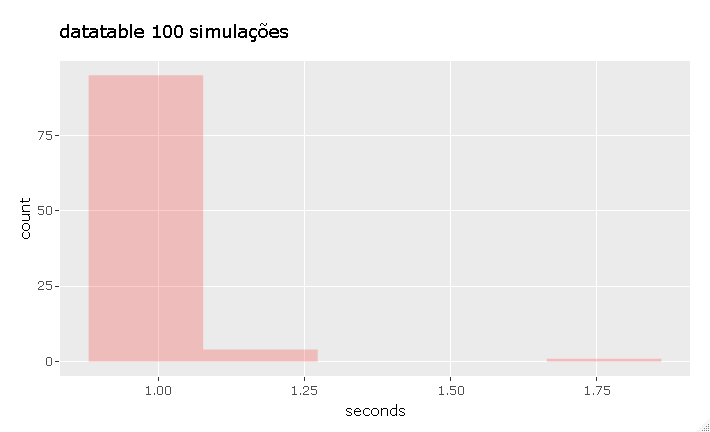
1 segundo com datatable e uma variação muito pequena desse tempo quando repetimos a ação várias vezes, já o dplyr leva de 2 à 4 segundos. A coisa fica pior para o dplyr se tivermos interesse num grupo maior e em mais resultados no summarize.
time_champs2 <- microbenchmark({
df3[,.(min_X3 = min(X3),
max_X3 = max(X3)),.(X1,X2)][order(X1,X2)]
}, times = 10L)
time_nonchamps2 <- microbenchmark({
df3 %>%
group_by(X1,X2) %>%
summarize(min_X3 = min(X3),
max_X3 = max(X3))
}, times = 10L)
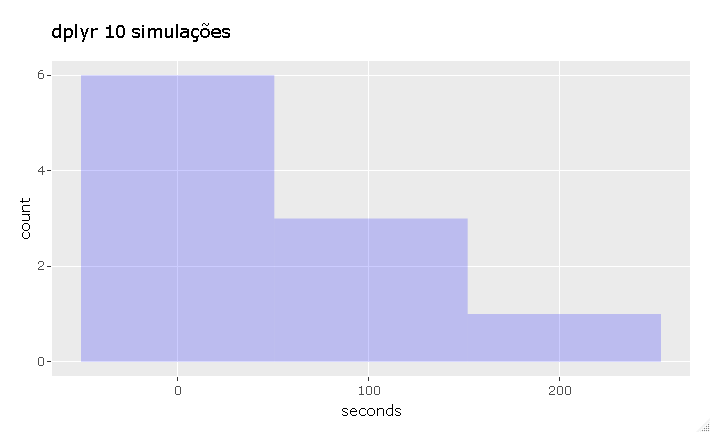
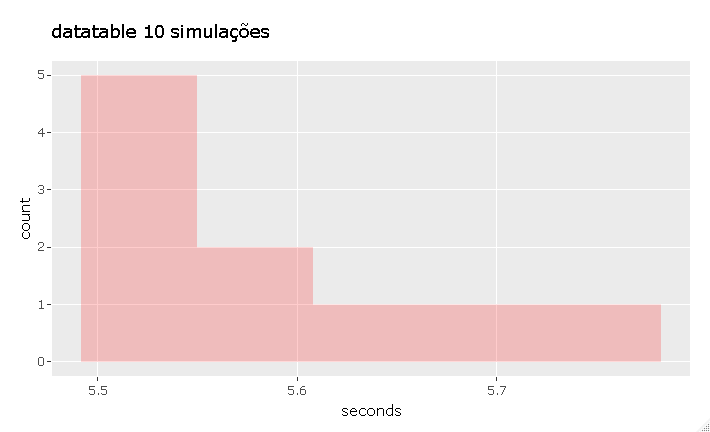
Aqui o dplyr levou de 28 à 230 segundos, contra 5.5 à 5.7 segundos do datatable.
Supondo que isso não seja suficiente para te convencer a dar uma chance ao datatable, vamos à um caso de join que tem uma solução muito eficaz e interessante em datatable.
d1<-data.table(v1=c("a","b","c","d","d","b","a","c","a","d","b","a"),
v2=(seq(1:12)),V3=rep(1:4,times=3))
head(d1)
v1 v2 V3
1: a 1 1
2: b 2 2
3: c 3 3
4: d 4 4
5: d 5 1
6: b 6 2
d2<-data.table(v1=c("a","b","c","d"),v3=c(3,2,1,4),v4=c("y","x","t","e"))
head(d2)
v1 v3 v4
1: a 3 y
2: b 2 x
3: c 1 t
4: d 4 e
O objetivo é colocar a informação da coluna v4 na base d1 para os casos onde os valores de v1 e v3 são equivalentes nas duas bases. No dplyr para resolver esse problema seria necessário usar um join, que copiaria d1 e ameaça explodir sua memória se d1 for grande. Com datatable, duas linhas resolvem o problema.
setkey(d1, v1, V3)
d1[d2, v4 := v4][]
# checando o resultado
v1 v2 V3 v4
1: a 1 1 <NA>
2: a 9 1 <NA>
3: a 7 3 y
4: a 12 4 <NA>
5: b 2 2 x
6: b 6 2 x
7: b 11 3 <NA>
8: c 3 3 <NA>
9: c 8 4 <NA>
10: d 5 1 <NA>
11: d 10 2 <NA>
12: d 4 4 e
Mágico, hein? Mas o que aconteceu?
- Primeiro indicamos as variáveis de d1 que serão consideradas chave.
- Em seguida temos d1[d2] que escrito dessa forma equivale a um right_join entre as bases utilizando as chaves definidas, mas ainda não é esse o objetivo, porque se o match não existir o que queremos ver em v4 é
NA. - := equivale ao mutate e vai adicionar a coluna de acordo com a referência, sendo assim as linhas sem match serão
NA. - Por fim, o [] no final serve para printar o resultado.
Fato curioso sobre este exemplo, é que ele é um caso real, que gerou ‘pergunta no stackoverflow’ uns anos atrás. A pessoa tinha uma base grande em mãos e já havia tentado loop, que se mostrou impraticável, depois partiu para merge e sqldf que não deram conta do problema.
Vale ressaltar que o data.table é tudo isso sim, mas ele não é para big data, ele é um last resort, se ficar impraticável com data.table é porque tem que partir uma ferramenta específica para manipular big data.
Últimas considerações
-
A inspiração principal para esse post veio quando eu comecei a perceber a frequência com que problemas do meu dia a dia, envolvendo manipulações pesadas, podiam ser resolvidos com datatable. Como inspiração secundária os créditos vão para ‘este post’ onde eu descobri que o jeito mais eficaz de agregar bases numa lista é com
data.table::rbindlist, além de pegar emprestado seu método de gerar aquela base enorme que uso no início, então créditos duplos a ele. -
Quer dar uma chance ao data.table, mas tá magoado com a sintaxe dele e/ou receoso de sair do dplyr que é tão organizado e intuitivo? ‘Aqui’ você encontra um de-para muito bem feito de ações importantes nas duas bibliotecas.
-
Para interessados, o datatable está sendo implementado para Python, isso deixa bem bacana a transição de uma linguagem para outra pois sair de
dplyrparapandasnão é trivial, mas se você usa data.table, vai manipular bases nas duas linguagens sem estresse. Mais detalhes ‘aqui -
Por último, porém talvez o mais importante para quem foi seduzido pelo datatable, ‘neste post do stackoverflow’ um camarada muito solícito faz uma análise detalhada da luta
data.table vs dplyr, levando em consideração os pilaresvelocidade,uso de memória,sintaxeefeatures.
E aí, partiu usar data.table?!